今回はDROMPA3のメイン機能であるマップリード分布の可視化を紹介します。
インストール
DROMPA3のインストール方法についてはこの記事を参照してください。
Genome table作成
DROMPA3の実行にはGenome tableファイルが必要になります。 Genome tableの作成は以下の記事を参照してください。
genome tableを作成する - Palmsonntagmorgen
遺伝子のアノテーション
可視化する際に必要になる遺伝子アノテーションデータ(refFlat形式)をUCSCのサイトからダウンロードします。
$ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/database/refFlat.txt.gz
$ gunzip refFlat.txt.gz # 解凍
parse2wig
今回は複数のChIPサンプルを可視化したいので、ENCODE projectにあるK562のヒストン修飾群を用います。 下記のコマンドでデータをダウンロード・生成しましょう。
$ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwHistone/wgEncodeUwHistoneK562H3k4me3StdAlnRep1.bam $ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwHistone/wgEncodeUwHistoneK562H3k27me3StdAlnRep1.bam $ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwHistone/wgEncodeUwHistoneK562H3k36me3StdAlnRep1.bam $ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwHistone/wgEncodeUwHistoneK562InputStdAlnRep1.bam $ parse2wig -i wgEncodeUwHistoneK562H3k4me3StdAlnRep1.bam -o H3K4me3 -gt genometable.txt -f BAM $ parse2wig -i wgEncodeUwHistoneK562H3k27me3StdAlnRep1.bam -o H3K27me3 -gt genometable.txt -f BAM $ parse2wig -i wgEncodeUwHistoneK562H3k36me3StdAlnRep1.bam -o H3K36me3 -gt genometable.txt -f BAM $ parse2wig -i wgEncodeUwHistoneK562InputStdAlnRep1.bam -o Input -gt genometable.txt -f BAM
drompa_draw
可視化にはdrompa_drawを使います。 "drompa_draw"とタイプすると、利用可能なコマンド一覧が出力されます。
$ drompa_draw Usage: drompa_draw [--version] <command> Command: PC_SHARP peak-calling (for sharp mode) PC_BROAD peak-calling (for broad mode) PC_ENRICH peak-calling (enrichment ratio) GV global-view visualization PD peak density ...
今回はPC_SHARPを使います。 "drompa_draw PC_SHARP"とコマンド含めてタイプすると、そのコマンドで利用可能なオプション一覧が表示されます。
$ drompa_draw PC_SHARP Usage: drompa_draw PC_SHARP [options] -p <output> -gt <genometable> -i <ChIP>,<Input>,<name> [-i <ChIP>,<Input>,<name> ...] <output>: Name of output files <genometable>: Tab-delimited file describing the name and length of each chromosome -i: specify ChIP data, Input data and name of ChIP sample (separated by ',', <Input> and <name> can be omitted) Options: (以下オプションが続く)
1サンプルの可視化
簡単のため、最初は1サンプルだけ使って可視化してみます。
$ drompa_draw PC_SHARP -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -p K562_H3K4me3 -gene refFlat.txt -gt genometable.txt
-i でChIP、Inputサンプルをカンマ区切りで指定するのはdrompa_peakcallと同じです。更にその後にコンマを入れて"H3K4me3"とタイプしていますが、これは出力図に表示されるサンプル名です。
-pで出力ファイル名、-geneで遺伝子アノテーションを指定します。
無事成功すると、以下のように全ゲノムを可視化したpdf(K562_H3K4me3.pdf)と、染色体別のpdfファイル(K562_H3K4me3_chr*.pdf)が作成されるでしょう。
$ ls
K562_H3K4me3.pdf K562_H3K4me3_chr14.pdf K562_H3K4me3_chr2.pdf K562_H3K4me3_chr5.pdf
K562_H3K4me3_chr1.pdf K562_H3K4me3_chr15.pdf K562_H3K4me3_chr20.pdf K562_H3K4me3_chr6.pdf
K562_H3K4me3_chr10.pdf K562_H3K4me3_chr16.pdf K562_H3K4me3_chr21.pdf K562_H3K4me3_chr7.pdf
K562_H3K4me3_chr11.pdf K562_H3K4me3_chr17.pdf K562_H3K4me3_chr22.pdf K562_H3K4me3_chr8.pdf
K562_H3K4me3_chr12.pdf K562_H3K4me3_chr18.pdf K562_H3K4me3_chr3.pdf K562_H3K4me3_chr9.pdf
K562_H3K4me3_chr13.pdf K562_H3K4me3_chr19.pdf K562_H3K4me3_chr4.pdf K562_H3K4me3_chrX.pdf
染色体別ファイルが必要ない時は-rmchrオプションを追加すると全ゲノムpdfのみ生成します。
また、例えば染色体1番のみで良い場合は、以下のように-chr 1と追加するとよいでしょう。
$ drompa_draw PC_SHARP -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -p K562_H3K4me3 -gene refFlat.txt -gt genometable.txt -chr 1 ... $ ls K562_H3K4me3.pdf K562_H3K4me3_chr1.pdf
K562_H3K4me3_chr1.pdf を開いてみましょう。
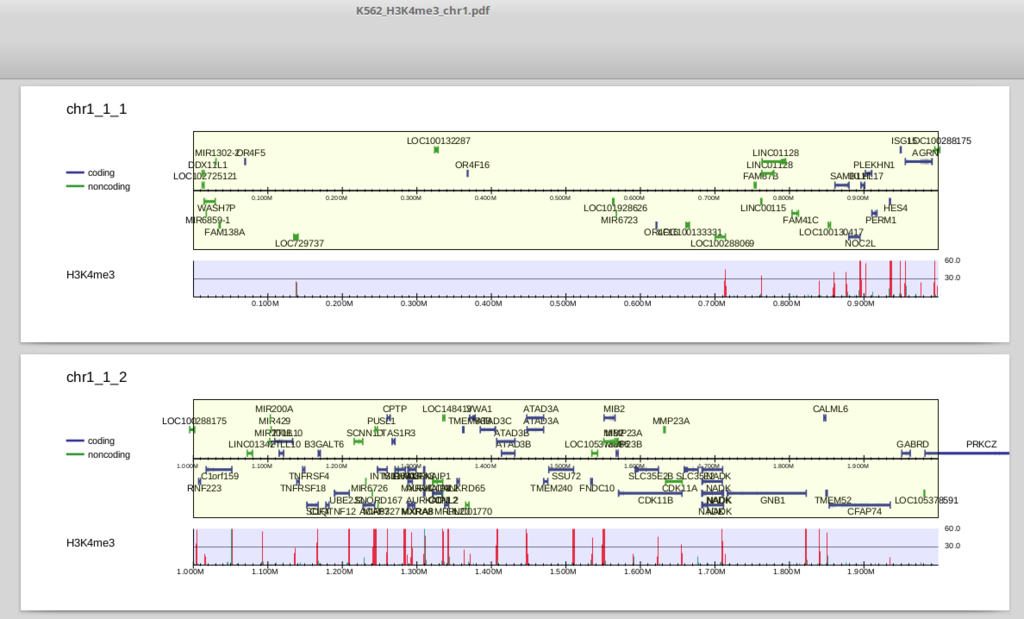
上部は遺伝子領域です。中央より上にあるものは順鎖、下にあるものは逆鎖です。refFlatデータはisoformを区別しますので、表示される遺伝子もtranscript単位になっています(ほとんど重なるものは省略されます)。
下部はリード分布で、ピーク領域(drompa_peakcallで抽出されるものと同一です)は赤、それ以外の領域は緑で表示されます(この図だとほとんど赤になっていますが)。
1段に表示されるゲノム幅(x軸)は-ls、y軸のスケールは-scale_tag、1ぺージ当たりに表示される段数は-lppで指定することができます。
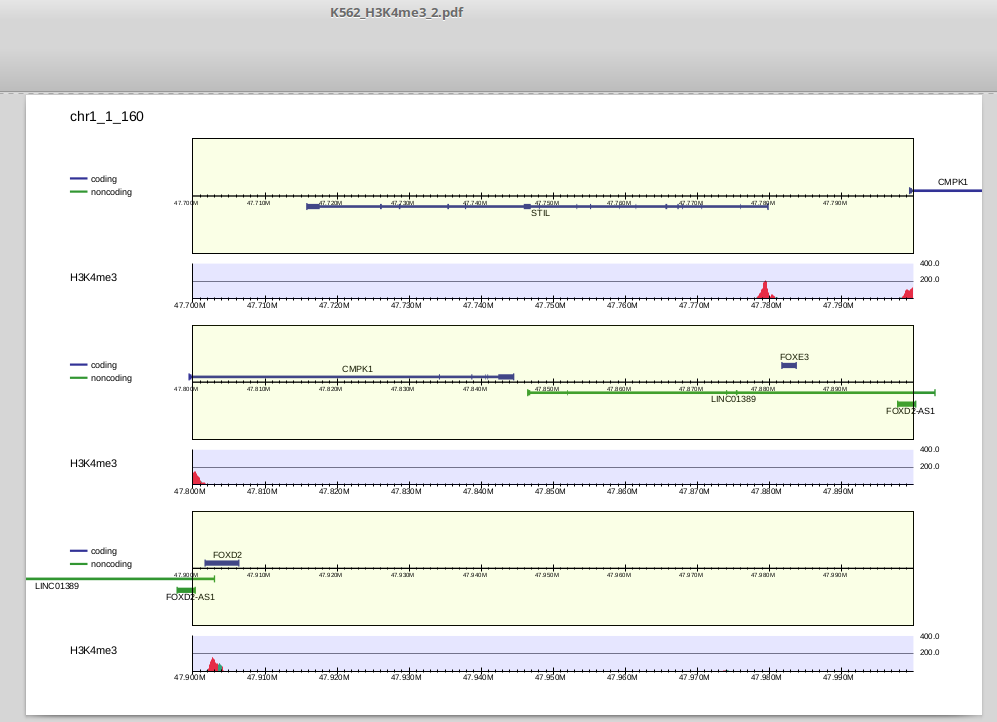
以下のようにパラメータを変えてみましょう。
$ drompa_draw PC_SHARP -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -p K562_H3K4me3_2 -gene refFlat.txt -gt genometable.txt -chr 1 \ -ls 100 -scale_tag 200 -lpp 3
出力は以下のようになります。
複数サンプルの可視化
4種類のヒストン修飾を並べて可視化してみましょう。
それぞれのサンプル(ChIP・Inputペア)を-iで指定します。コマンドは以下のようになります。
$ drompa_draw PC_SHARP \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -p K562 -gene refFlat.txt -gt genometable.txt -chr 1 -lpp 2
-iで指定した順番に並べて可視化されます。
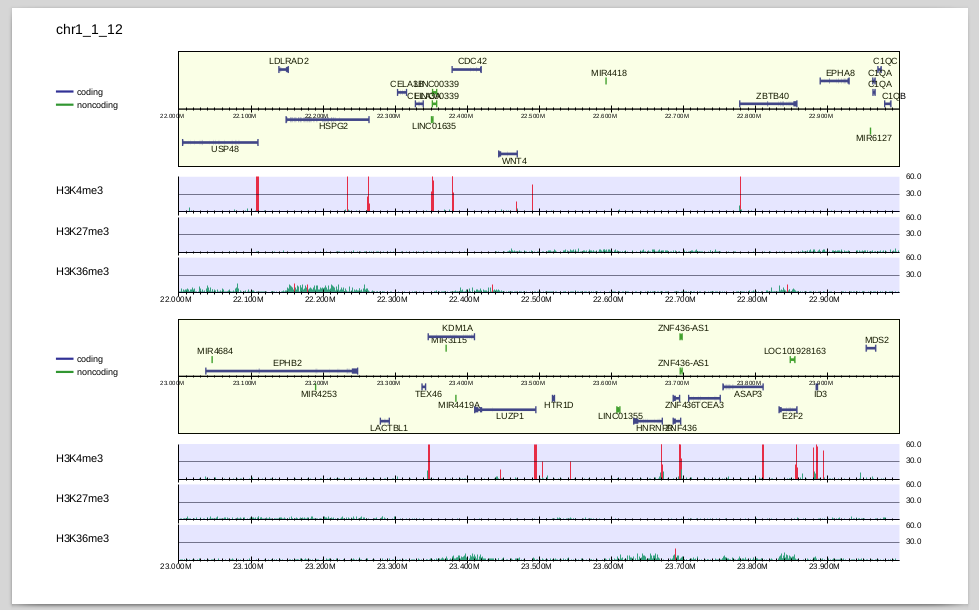
H3K27me3, H3K36me3はbroad peakであり、H3K4me3に比べるとリードの濃縮が弱いので、縦軸が同じスケールだとわかりづらいですね。 そこで、個別にスケールを変えてみましょう。
$ drompa_draw PC_SHARP \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3,,,200 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3,,,10 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3,,,10 \ -p K562 -gene refFlat.txt -gt genometable.txt -chr 1 -lpp 2
-i の項目で最後に指定した数字(200,10,10)が、それぞれのサンプルのy軸のスケールになります。ここで指定したスケールは-scale_tagよりも優先されます。
何故カンマが3つ続くのかは、、次回以降に解説しますので、ここでは置いておきます。
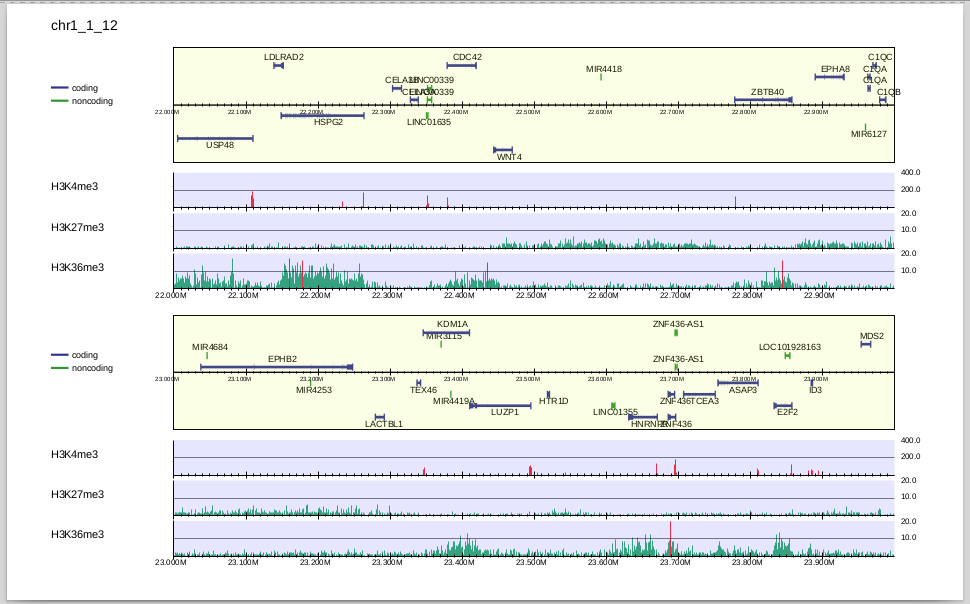
y軸のスケールが変わり、H3K27me3, H3K36me3の濃縮が見えるようになりました。 H3K36me3はtranscribed stateのマーカー、H3K27me3はsuppressive stateのマーカーで、ゲノム上で排他的に修飾が入ることが知られていますが、それがこの図からもわかります。
以上がDROMPA3での最も基本的な可視化です。これからしばらくは可視化機能について解説していこうと思います。