今回はDROMPAを用いたヒートマップの描画について説明します。
HEATMAPコマンド(TSS周辺)
前回の記事では、DROMPAを用いたリードプロファイルの描画について解説しました。
使ったコマンドは以下のようなものです。(詳しくは前回の記事を参照してください)
drompa_draw PROFILE \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -p profile.K562 -gene refFlat.txt -gt genometable.txt
ヒートマップの描画はプロファイルと非常に似ています。 上のコマンドを以下のように少し変えるだけでヒートマップのコマンドになります。
drompa_draw HEATMAP \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -p K562 -gene refFlat.txt -gt genometable.txt -png
変えた部分は、PROFILEをHEATMAPに変更したことと、ファイル名(-pオプション)、-pngオプションを付け加えたことです。
デフォルトではpdfでファイルが出力されますが、ヒートマップをpdfで出力するとしばしばファイルサイズが大きくなりすぎる&そこまで高解像度は必要ないため、-pngオプションを付けて.pngで出力させた方が扱いやすくなります。
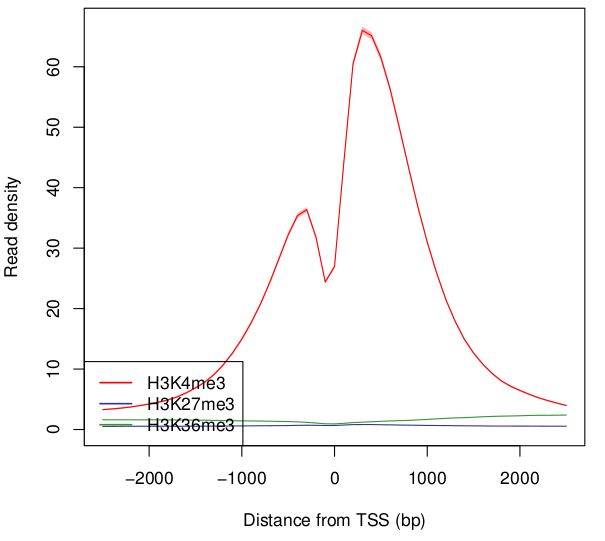
PROFILEコマンドと同様、デフォルトでは入力された遺伝子の転写開始点周辺5kbを可視化します。
出力されたK562.heatmap.pngを開いてみましょう。

全遺伝子をヒートマップ化しているのでかなり縦長になります。ここに表示したのは一部です。
H3K4me3はTSSまわりに濃縮が見られる一方、H3K27me3, H3K36me3は強い濃縮が見られないことがわかります。
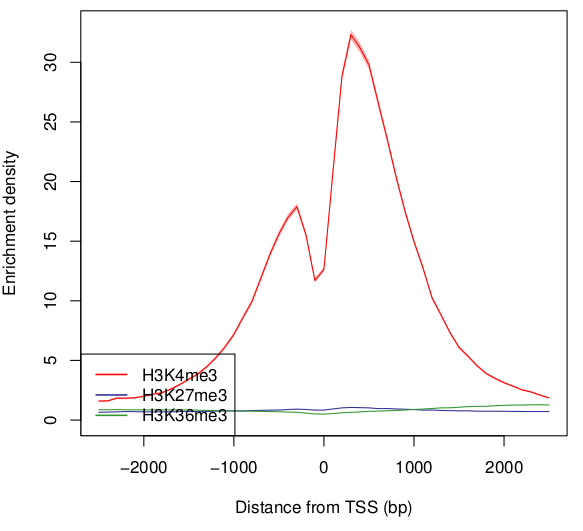
しかし、色のスケールを変えることで違いが出るかもしれません。
-scale_tag 5 オプションを追加して、色のMAX値を5に変更してみましょう。
drompa_draw HEATMAP \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -p K562-2 -gene refFlat.txt -gt genometable.txt -png -scale_tag 5

どうやら、H3K27me3よりはH3K36me3 の方がTSSまわりに濃縮があるようです。
HEATMAPコマンド(ピーク周辺、ソートなし)
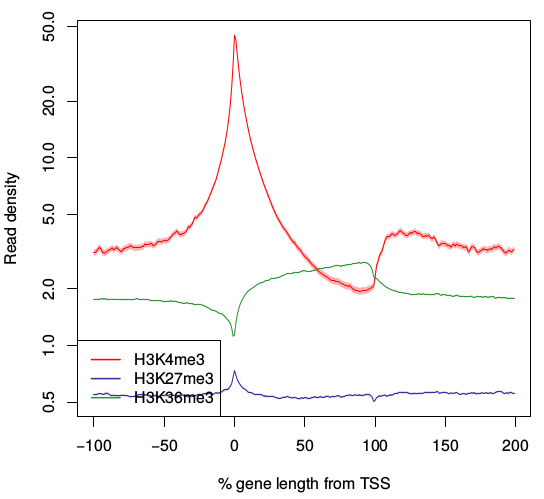
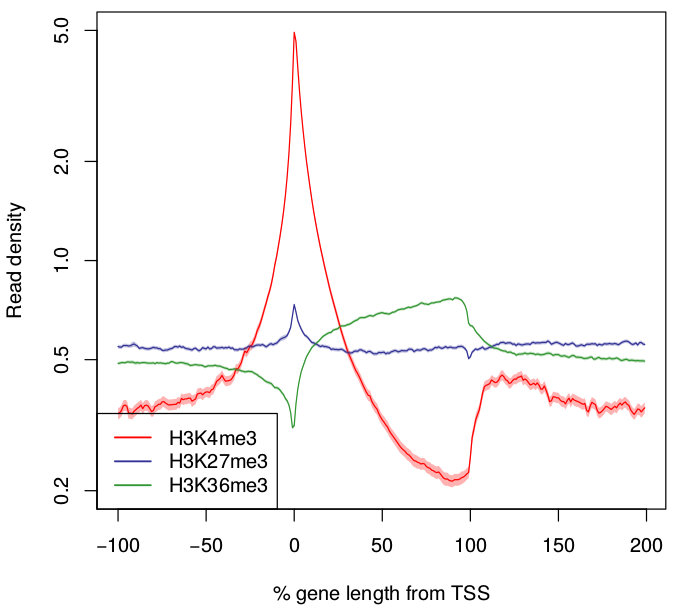
HEATMAPコマンドもPROFILEコマンド同様、ピーク周辺を可視化することができます。 また、複数のピークファイルを入力とすることができます。
ここではENCODEのオープンクロマチン領域を利用してみます。まずデータをダウンロードしましょう。
wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeOpenChromSynth/wgEncodeOpenChromSynthK562Pk.bed.gz wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeOpenChromSynth/wgEncodeOpenChromSynthHepg2Pk.bed.gz # 解凍 gunzip wgEncodeOpenChromSynthK562Pk.bed.gz gunzip wgEncodeOpenChromSynthHepg2Pk.bed.gz # ピーク数が多いので、最初の1000行を抽出 head -n 1000 wgEncodeOpenChromSynthHepg2Pk.bed > wgEncodeOpenChromSynthHepg2Pk.1000.bed head -n 1000 wgEncodeOpenChromSynthHepg2Pk.bed > wgEncodeOpenChromSynthHepg2Pk.1000.bed
DROMPAコマンドは以下のようになります。
-bed コマンドは、ファイル名とラベル(出力図y軸に表示されるもの)をコンマで区切って指定します。
drompa_draw HEATMAP \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -bed wgEncodeOpenChromSynthK562Pk.1000.bed,open_K562 \ -bed wgEncodeOpenChromSynthHepg2Pk.1000.bed,open_HepG2 \ -p K562-3 -ptype 4 -gt genometable.txt -png

2つのbedファイルで指定された領域が線で区切られて出力されました。
HEATMAPコマンド(ピーク周辺、ソートあり)
y軸はデフォルトでは入力ファイルの通りの順で出力されますが、
以下のように-hmsort オプションをつけると、ピーク強度でソートされます。
drompa_draw HEATMAP \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -bed wgEncodeOpenChromSynthK562Pk.1000.bed,open_K562 \ -bed wgEncodeOpenChromSynthHepg2Pk.1000.bed,open_HepG2 \ -p K562-4 -ptype 4 -gt genometable.txt -png -hmsort 1

-hmsort 1とすると、1番目のサンプル(ここではH3K4me3)のピーク強度に基づいてソートされます。また、中心のリード数が0のサイトは除外されます。
こうしてみると、K562のオープンクロマチンサイトだけでなく、HepG2のオープンクロマチンサイトにもH3K4me3の濃縮が見られます。両方の細胞で共通するオープンクロマチンサイトなのかもしれません。
ここではH3K4me3にしか強い濃縮が見られませんが、共起するピークとそうでないピークに分けてベン図と組み合わせてヒートマップを使うと効果的にピークの濃縮を可視化することができます。
ヒートマップの特徴
PROFILEコマンドでは平均値(と信頼区間)を可視化しますが、この場合、全入力領域のうち全てに濃縮が出ているのか、あるいは半々程度にばらついているのか区別ができません。ヒートマップを使うと、全体のどのくらいの割合に濃縮が出ているのか、サンプル間で共通性・排他性があるのかなどを視覚的にとらえることができます。
一方、ヒートマップは統計的な処理をしているわけではないので、通常のリードプロファイルと同様、特徴的な領域を直感的に例示する場合などには有効ですが、全ゲノムを定量的に分析するような用途には使えません。