DROMPA3解説その3はピーク抽出(peak calling)です。ピーク抽出とは、ゲノムからreadが有意に濃縮している箇所を網羅的に同定する作業です。
インストール
DROMPA3でのピーク抽出は、drompa_peakcall を使います。
DROMPA3のインストール方法についてはこの記事を参照してください。
単にdrompa_peakcallとタイプすると、以下のヘルプが表示されます。
$ drompa_peakcall Usage: drompa_peakcall [--version] <command> Command: PC_SHARP peak-calling (for sharp mode) PC_BROAD peak-calling (for broad mode) PC_ENRICH peak-calling (enrichment ratio)
Genome table作成
DROMPA3の実行にはGenome tableファイルが必要になります。 Genome tableの作成は以下の記事を参照してください。
genome tableを作成する - Palmsonntagmorgen
parse2wig
前回のエントリでparse2wigを用いて作成したファイルを使います。 とりあえず下記のコマンドでデータは生成できます。
$ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwTfbs/wgEncodeUwTfbsHelas3CtcfStdAlnRep1.bam $ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwTfbs/wgEncodeUwTfbsHelas3InputStdAlnRep1.bam $ parse2wig -i wgEncodeUwTfbsHelas3CtcfStdAlnRep1.bam -o Ctcf -gt genometable.txt -f BAM $ parse2wig -i wgEncodeUwTfbsHelas3InputStdAlnRep1.bam -o Input -gt genometable.txt -f BAM
ピーク抽出
drompa_peakcall は以下の3種類の内部コマンドを持ちます。
- PC_SHARP: 転写因子などの鋭いピーク(sharp peak)用
- PC_BROAD: 一部のヒストン修飾など広範囲に分布するピーク(broad peak)用
- PC_ENRICH: 有意差検定を用いずにシンプルにChIP/Input比のみを考慮するコマンド
ちなみにこの3つのコマンドはデフォルトパラメータが異なるだけで、基本的は一緒だと思ってもらって構いません。ここではPC_SHARPを使いましょう。
下記のようにコマンド含めてタイプすると、オプション一覧が表示されます。
$ drompa_peakcall PC_SHARP please specify output prefix. Usage: drompa_peakcall COMMAND [options] -p <output> -gt <genometable> -i <ChIP>,<Input> <ChIP> ChIP sample <Input> Input (control) sample <output>: Name of output files <genometable>: Tab-delimited file describing the name and length of each chromosome Options: (以下オプションが続く)
前回生成したファイルを指定して、CTCFのピークを抽出してみましょう。
$ drompa_peakcall PC_SHARP -i parse2wigdir/Ctcf,parse2wigdir/Input -gt genometable.txt -p CTCF
-iは入力とするデータで、ChIPファイルと対応するInputファイルをカンマで区切って指定します。-gtはGenome table、-pは出力ファイル名です。
このコマンドを実行すると、以下のようなログが標準出力に現れます。
======== PC_SHARP ======== drompa_peakcall version 3.3.0 output prefix: CTCF genometable: genometable.txt ChIP: parse2wigdir/Ctcf Input: parse2wigdir/Input Input format: BINARY binsize: 100 smoothing width: 500 bp Peak intensity threshold: 0.00 ChIP/Input normalization: TOTALREAD Enrichment threshold: 0.00 p-value threshold (internal, -log10): 4.00e+00 p-value threshold (enrichment, -log10): 4.00e+00 q-value threshold: 1.00e-03 ====================================== sample1: genome: ChIP read=4757329, Input read=14682144, ChIP/Input = 0.32 Peak-calling: chr1..chr2..chr3..chr4..chr5..chr6..chr7..chr8..chr9..chr10..chr11..chr12..chr13..chr14..chr15..chr16..chr17..chr18..chr19..chr20..chr21..chr22..chrX..done. output peaklist...done.
コマンド実行終了後、カレントディレクトリにCTCF.bed と CTCF.xls という2種類のピークリストが生成されます。この2つのファイルの内容は同一ですが、CTCF.bedはBED形式、CTCF.xlsはより詳細な情報を含むcsv形式という違いがあります。
ピーク抽出の閾値
DROMPA3は以下の五種類のピーク抽出閾値パラメータを持ちます。
threshold:
-pthre_internal <double>: p-value threshold for ChIP internal (Poisson, default < 1.0e-04)
-pthre_enrich <double>: p-value threshold for ChIP/Input enrichment (binomial, default < 1.0e-04)
-ethre <double>: IP/Input enrichment threshold (default: 2 for PC_ENRICH, 0 for the others)
-ipm <double>: read intensity threshold of peak summit (default: 0)
-qthre <double>: q-value threshold (Bonferroni-Hochberg, default < 0.001)
このうち、-ethre (ChIP/Input比の閾値)、-ipm(ピークの高さの閾値)はPC_SHARPモードではデフォルトでは用いないので、残り3つのパラメータが閾値となります。
有意差検定
DROMPA3は内部で以下の2種類の有意差検定を行います。この2段階検定の方法はPeakSeqのアルゴリズムに基づきます*1。
- ChIP internal: ChIPサンプルにおいて、周辺に対してそのウィンドウにreadが有意に濃縮しているか。(負の二項検定)
- ChIP/Input enrichment: そのウィンドウにおいで、Inputに対してChIPサンプルのreadが有意に濃縮しているか(二項検定)
図で表すと以下のようになります。
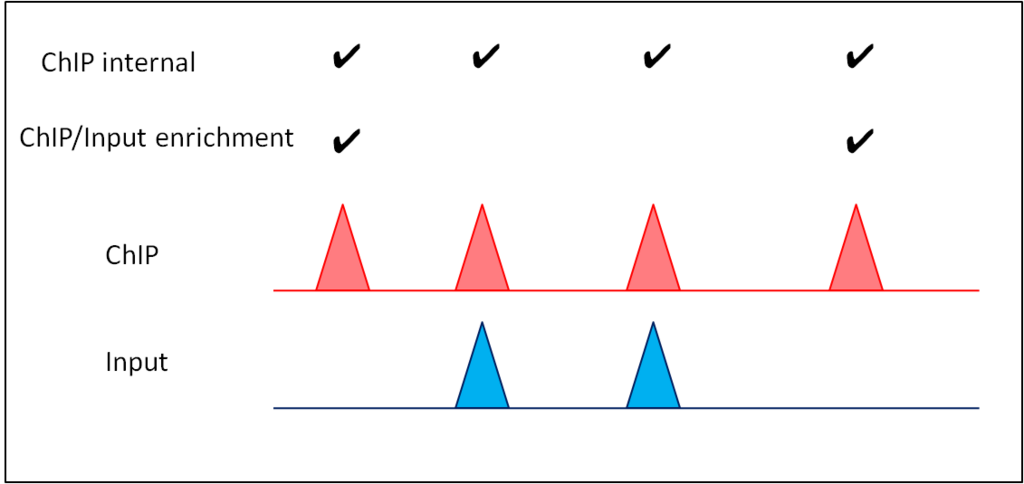
ChIP internalの検定ではピーク様の濃縮領域を候補領域として全て抽出し、そのうちChIP/Input比が有意であるもの、すなわちInputでは濃縮していない領域のみを抽出します。通常、ChIP/Input enrichmentの閾値の値がピーク数にクリティカルに効いてきますので、ピーク抽出の閾値を変えてみたい場合は、-pthre_enrich オプションの値を色々と変えてみると良いでしょう。
なお、drompa_peakcall はInputサンプル無しでもピーク抽出可能ですが、その場合はChIP/Input enrichmentの検定は省略され、ChIP internalで有意とされた領域全てをピークとして出力します。
このように抽出されたピークリストに対し、さらにBenjamini-Hochberg法による多重検定の補正*2を行います(-qthre)。なお経験的には、ピーク数が数千以上の場合はこの閾値はほとんど関係ありません。。
オプション
-binsize: デフォルトでは100bpですが、異なるサイズにすることができます。その場合、parse2wigでそのサイズのデータをあらかじめ生成している必要があります。-sm: スムージングをかける幅です。デフォルトでは500bpです。スムージングをかけると細かいノイズの影響を低減することができますが、ピークの幅が広くなるため、モチーフ解析などをしたい場合は-sm 0としてスムージングをオフにすると良いでしょう。-norm: デフォルトでは、有意差検定を行う際にChIP/Input比が1になるようにread数の正規化を行います。-norm 0とすると正規化をオフにします。-includeYM: デフォルトではY染色体とミトコンドリアのピークは無視されますが、このオプションを追加すると出力されるようになります。
他にもいくつかオプションがあるのですが、それはまた別エントリで解説したいと思います。毎回エントリが長くなり過ぎですね…もうすこしコンパクトにしたいのですが。
とりあえず可視化まではこのまま進もうと思います。
*1:Rozowsky J, Euskirchen G, Auerbach RK, et al. PeakSeq enables systematic scoring of ChIP-seq experiments relative to controls. Nat Biotechnol 2009;27:66–75.
*2:Benjamini-Hochberg法についてはこちらのエントリが参考になります:Benjamini-Hochberg法の原理 - ほくそ笑む