DROMPA3を用いたリードプロファイルの描画です。
ここでプロファイルと呼んでいるものは、遺伝子回り、あるいはピーク回りにおけるマップリードの平均分布のことで、aggregation plotと呼ばれることもあります。
全遺伝子や全ピークの平均値として見ることにより、通常のピーク抽出では得られない微小なリードの濃縮を捉えたり、発現遺伝子群と非発現遺伝子群の間でのリード濃縮の違いを可視化することができます。
インストール
DROMPA3のインストール方法はこの記事を参照してください。
PROFILEコマンドは内部でRスクリプトを実行しますので、Rが必要になります。
Genome table作成
DROMPA3の実行にはGenome tableファイルが必要になります。 Genome tableの作成は以下の記事を参照してください。
genome tableを作成する - Palmsonntagmorgen
遺伝子アノテーション
可視化する際に必要になる遺伝子アノテーションデータ(refFlat形式)をUCSCのサイトからダウンロードします。
$ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/database/refFlat.txt.gz
$ gunzip refFlat.txt.gz # 解凍
parse2wig
今回もENCODE projectにあるK562のヒストン修飾群、遺伝子アノテーションを用います。
$ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwHistone/wgEncodeUwHistoneK562H3k4me3StdAlnRep1.bam $ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwHistone/wgEncodeUwHistoneK562H3k27me3StdAlnRep1.bam $ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwHistone/wgEncodeUwHistoneK562H3k36me3StdAlnRep1.bam $ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/encodeDCC/wgEncodeUwHistone/wgEncodeUwHistoneK562InputStdAlnRep1.bam $ parse2wig -i wgEncodeUwHistoneK562H3k4me3StdAlnRep1.bam -o H3K4me3 -gt genometable.txt -f BAM $ parse2wig -i wgEncodeUwHistoneK562H3k27me3StdAlnRep1.bam -o H3K27me3 -gt genometable.txt -f BAM $ parse2wig -i wgEncodeUwHistoneK562H3k36me3StdAlnRep1.bam -o H3K36me3 -gt genometable.txt -f BAM $ parse2wig -i wgEncodeUwHistoneK562InputStdAlnRep1.bam -o Input -gt genometable.txt -f BAM
PROFILEコマンド
drompa_draw PROFILE とタイプするとヘルプが表示されます。
$ drompa_draw PROFILE Usage: drompa_draw PROFILE [options] -p <output> -gt <genometable> -i <ChIP>,<Input>,<name> [-i <ChIP>,<Input>,<name> ...] <output>: Name of output files <genometable>: Tab-delimited file describing the name and length of each chromosome -i: specify ChIP data, Input data and name of ChIP sample (separated by ',', <Input> and <name> can be omitted) Options: (以下オプションが続く)
まずはデフォルトで実行してみましょう。コマンドは以下のようになります。
drompa_draw PROFILE \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -p profile.K562 -gene refFlat.txt -gt genometable.txt
このコマンドにより以下のprofile.K562.pdfが生成されます。
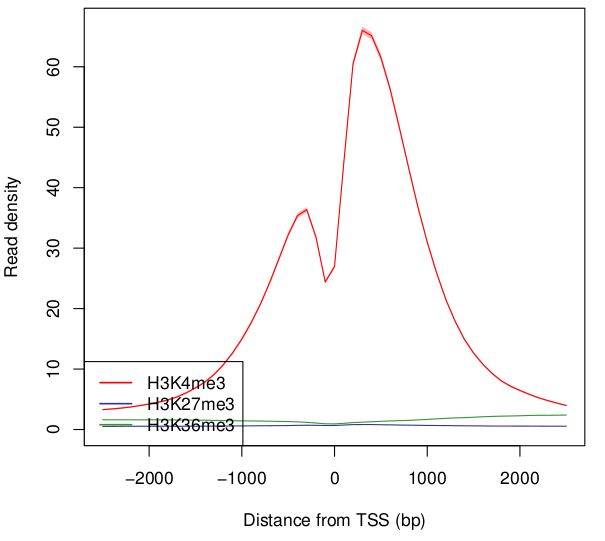
このプロファイルは遺伝子の転写開始点(TSS)周辺5kbp (±2.5kbp) の平均マップリード数を可視化したものです。ラインが各サンプルの平均リード分布、陰になっている領域が95%信頼区間を表しています。
この図を見ると、H3K4me3はTSS周辺にリードの濃縮がみられる一方、H3K27me3とH3K36me3は濃縮がみられないことがわかります。
オプション
-cwオプションをつけると可視化する周辺長の長さを変えることができます。±5kbpのプロファイルを生成するには以下のようにします。
drompa_draw PROFILE \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -p profile.K562 -gene refFlat.txt -gt genometable.txt -cw 5000

デフォルトではChIPサンプルのリードの平均値を描画しますが、-stype 1 をつけるとChIP/InputのEnrichmentの平均値を描画します。
drompa_draw PROFILE \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -p profile.K562 -gene refFlat.txt -gt genometable.txt -stype 1
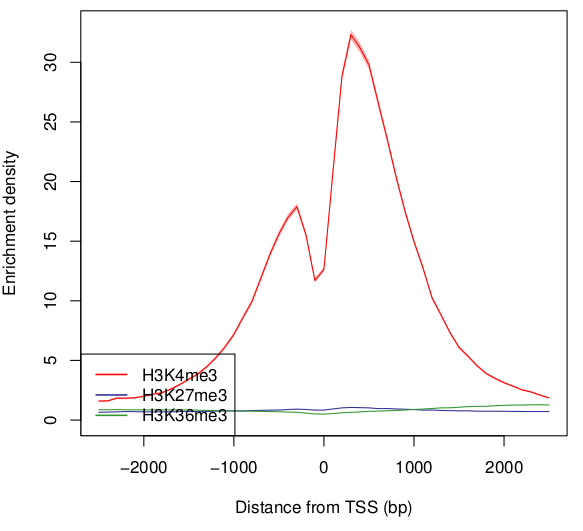
この例だとChIP readの時との差がわかりにくいですが、broad markや酵母の複製解析などでは差が顕著になることもあります。
(これは各TSSのChIP/Input の平均値であり、ChIP平均/Input平均のEnrichmentではないことに注意。)
遺伝子全長プロファイル
次に、遺伝子全長を100分割したプロファイルを作ってみましょう。 -ptype 3をつけて実行します。
drompa_draw PROFILE -ptype 3 \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -p profile.K562.ptype3 -gene refFlat.txt -gt genometable.txt
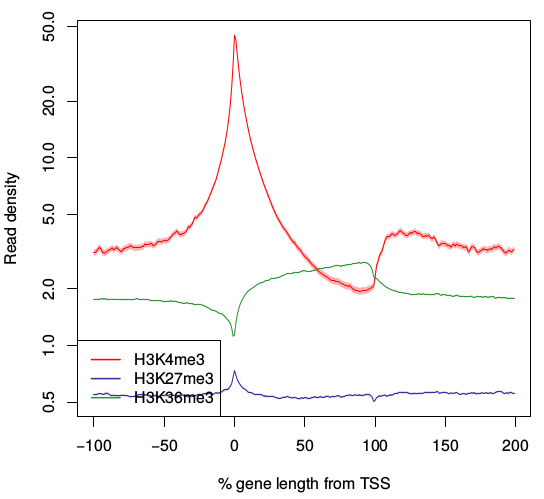
このプロファイルは各遺伝子について長さを一定に正規化したうえで遺伝子長を100分割し、遺伝子内(gene body)でどのように濃縮しているかを見たものです。
x軸の0がTSS、100がTES(転写終了点)で、その両側に遺伝子長と同じ長さだけ伸長した領域について計測します。
(従って遺伝子ごとに計測している領域の幅が異なることに注意)
100分割した1つのウィンドウがwigファイルの1bin (100bp)より大きい場合、その領域内の全binの平均値を出力します。
TSS周辺の可視化ではほとんど平らだったH3K36me3ですが、遺伝子全体で見るとgene bodyに広くゆるやかな濃縮が広がっていることがわかります。H3K36me3は転写中のgene bodyに入る修飾ですので、これは妥当な結果と言えます。
H3K27me3の方はTSS周辺に微小な濃縮があるようですが、こちらは意味があるかどうか微妙なところです。
領域内でのリード数正規化
3つのサンプル間で縦軸がずれている(平均リード値が異なる)のが気になる場合は、-ntype 1オプションを追加すると、観測している領域内の総リード数でサンプルごとに正規化します。
drompa_draw PROFILE -ptype 3 -ntype 1 \ -i parse2wigdir/H3K4me3,parse2wigdir/Input,H3K4me3 \ -i parse2wigdir/H3K27me3,parse2wigdir/Input,H3K27me3 \ -i parse2wigdir/H3K36me3,parse2wigdir/Input,H3K36me3 \ -p profile.K562.ptype3 -gene refFlat.txt -gt genometable.txt
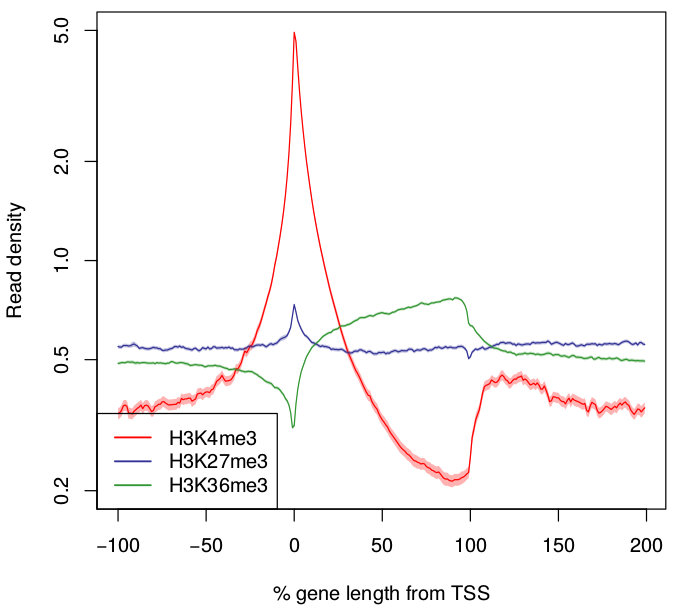
完全ではありませんが、遺伝子外領域の高さがだいたい同じになりました。
まとめ
遺伝子全長の可視化は、H3K36me3のようにgene bodyに入るヒストン修飾や、RNA pol2のようにgene bodyに広く分布する(伸長する)タンパク質のChIP-seqを可視化するのに有効です。
今回はTSS周辺と遺伝子全長について可視化しましたが、-ptype 2でTES周辺、-ptype 4で指定したピークリスト周辺の濃縮を可視化することができます。
また、同時に生成されるRスクリプトprofile.K562.R を編集の後以下のように実行することで、ラベルやy軸のスケールを変更したpdfファイルを再生成することも可能です。
R --vanilla < profile.K562.R
プロファイルの場合全体の何割で濃縮が入っているかはわかりませんが、信頼区間の幅を見ることである程度推測することが可能です。