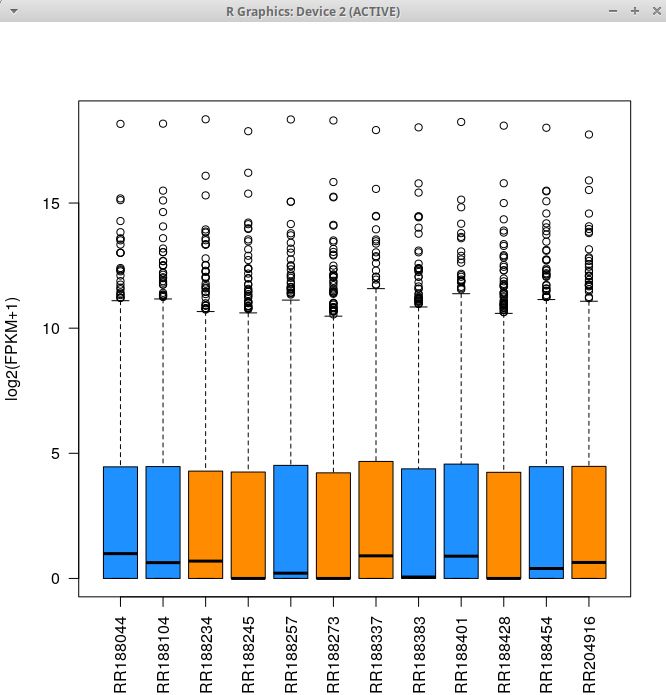
RNA-seq解析の記事を書こうとしたらgtfファイルの部分が長くなり過ぎたので単独記事にしました。
Gene annotation
既知遺伝子の情報を記載するファイルにはいくつかの形式があり、gtf形式はそのひとつです。 よく似たgff形式というものもありますが、微妙に異なります。
gtf形式の詳細については以下の記事を参考にしてください(丸投げ)。
GTFファイルのダウンロード
遺伝子情報はダウンロード元のデータベースによってIDや情報の詳細度がかなり違います。 ここでは私がよく使うUCSC genome browserとEnsemblからダウンロードしてみたいと思います。
UCSC genome browser
Table browserで遺伝子情報を指定し、"get output" ボタンを押します。RefseqのHuman build hg38 であれば、以下のようになります。
Ensembl
Ensemblは各種データを↓のFTP siteからダウンロード可能です。
http://asia.ensembl.org/info/data/ftp/index.html
Human のGRCh38 (hg38に対応)データであれば、以下のコマンドでダウンロードできます。
$ wget ftp://ftp.ensembl.org/pub/release-94/gtf/homo_sapiens/Homo_sapiens.GRCh38.94.gtf.gz
scaffoldなどの遺伝子情報を含んだgtfファイルも別にありますが、通常は使いません。
ダウンロードした二つのファイルを解凍後、サイズを比較してみましょう。
$ gunzip *.gtf.gz $ ls -lh *.gtf -rwxrwxrwx 1 rnakato rnakato 1.1G Dec 13 11:17 Homo_sapiens.GRCh38.94.gtf -rwxrwxrwx 1 rnakato rnakato 50M Dec 13 11:13 UCSC.hg38.gtf
Ensemblの方がファイルサイズが圧倒的に大きいですね。解凍すると1Gbを超えています。Ensemblの方がprocessed transcriptやpseudogeneなどの情報が多く、アノテーションも詳細に記述されていますので、遺伝子情報をもとに詳細な解析をしたい場合はEnsemblを使う方が良いと思います。
染色体名の違い
UCSC と Ensembl はgene IDが違いますが、染色体名も違います。UCSCの方は 1番染色体は"chr1"となっていますが、Ensemblは単に"1"となっています。UCSCのgenomeファイルを用いて解析している場合、この違いを考慮しないツールであれば「染色体名が異なる」とエラーになり、これが地味に面倒です。
ゲノムの染色体名の方をEnsemblに揃えるやり方もありますが、ここではEnsemblの遺伝子名にchrを追加する方法を試してみましょう。
修正前
$ head Homo_sapiens.GRCh38.94.gtf #!genome-build GRCh38.p12 #!genome-version GRCh38 #!genome-date 2013-12 #!genome-build-accession NCBI:GCA_000001405.27 #!genebuild-last-updated 2018-07 1 havana gene 11869 14409 . + . gene_id "ENSG00000223972"; gene_version "5"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; 1 havana transcript 11869 14409 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000456328"; transcript_version "2"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-202"; transcript_source "havana"; transcript_biotype "processed_transcript"; tag "basic"; transcript_support_level "1"; 1 havana exon 11869 12227 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000456328"; transcript_version "2"; exon_number "1"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-202"; transcript_source "havana"; transcript_biotype "processed_transcript"; exon_id "ENSE00002234944"; exon_version "1"; tag "basic"; transcript_support_level "1"; 1 havana exon 12613 12721 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000456328"; transcript_version "2"; exon_number "2"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-202"; transcript_source "havana"; transcript_biotype "processed_transcript"; exon_id "ENSE00003582793"; exon_version "1"; tag "basic"; transcript_support_level "1"; 1 havana exon 13221 14409 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000456328"; transcript_version "2"; exon_number "3"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-202"; transcript_source "havana"; transcript_biotype "processed_transcript"; exon_id "ENSE00002312635"; exon_version "1"; tag "basic"; transcript_support_level "1";
冒頭が染色体名になっているので、行頭にchrを付加するだけでよいですね。これは以下のようなワンライナーで実行可能です。
# 冒頭のコメントアウト文を除外し、各行の行頭にchrを追加して出力 $ grep -v \# Homo_sapiens.GRCh38.94.gtf | awk '{print "chr" $0 }' > Homo_sapiens.GRCh38.94.addchr.gtf # 内容確認 $ head Homo_sapiens.GRCh38.94.addchr.gtf head Homo_sapiens.GRCh38.94.addchr.gtf chr1 havana gene 11869 14409 . + . gene_id "ENSG00000223972"; gene_version "5"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; chr1 havana transcript 11869 14409 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000456328"; transcript_version "2"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-202"; transcript_source "havana"; transcript_biotype "processed_transcript"; tag "basic"; transcript_support_level "1"; chr1 havana exon 11869 12227 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000456328"; transcript_version "2"; exon_number "1"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-202"; transcript_source "havana"; transcript_biotype "processed_transcript"; exon_id "ENSE00002234944"; exon_version "1"; tag "basic"; transcript_support_level "1"; chr1 havana exon 12613 12721 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000456328"; transcript_version "2"; exon_number "2"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-202"; transcript_source "havana"; transcript_biotype "processed_transcript"; exon_id "ENSE00003582793"; exon_version "1"; tag "basic"; transcript_support_level "1"; chr1 havana exon 13221 14409 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000456328"; transcript_version "2"; exon_number "3"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-202"; transcript_source "havana"; transcript_biotype "processed_transcript"; exon_id "ENSE00002312635"; exon_version "1"; tag "basic"; transcript_support_level "1"; chr1 havana transcript 12010 13670 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000450305"; transcript_version "2"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-201"; transcript_source "havana"; transcript_biotype "transcribed_unprocessed_pseudogene"; tag "basic"; transcript_support_level "NA"; chr1 havana exon 12010 12057 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000450305"; transcript_version "2"; exon_number "1"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-201"; transcript_source "havana"; transcript_biotype "transcribed_unprocessed_pseudogene"; exon_id "ENSE00001948541"; exon_version "1"; tag "basic"; transcript_support_level "NA"; chr1 havana exon 12179 12227 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000450305"; transcript_version "2"; exon_number "2"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-201"; transcript_source "havana"; transcript_biotype "transcribed_unprocessed_pseudogene"; exon_id "ENSE00001671638"; exon_version "2"; tag "basic"; transcript_support_level "NA"; chr1 havana exon 12613 12697 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000450305"; transcript_version "2"; exon_number "3"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-201"; transcript_source "havana"; transcript_biotype "transcribed_unprocessed_pseudogene"; exon_id "ENSE00001758273"; exon_version "2"; tag "basic"; transcript_support_level "NA"; chr1 havana exon 12975 13052 . + . gene_id "ENSG00000223972"; gene_version "5"; transcript_id "ENST00000450305"; transcript_version "2"; exon_number "4"; gene_name "DDX11L1"; gene_source "havana"; gene_biotype "transcribed_unprocessed_pseudogene"; transcript_name "DDX11L1-201"; transcript_source "havana"; transcript_biotype "transcribed_unprocessed_pseudogene"; exon_id "ENSE00001799933"; exon_version "2"; tag "basic"; transcript_support_level "NA";
無事追加されました。
冒頭のコメント文を除外したくない場合は後から付け足すか、最初のコマンドでgrep部分を使わずに出力してください(後で手作業で#前のchrを取り除く)。