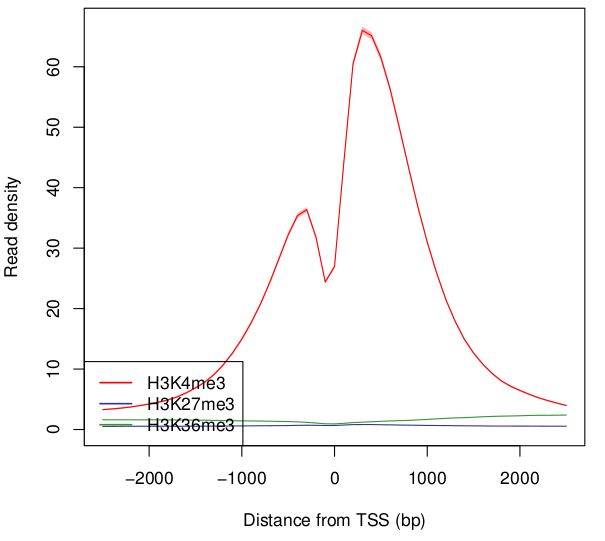
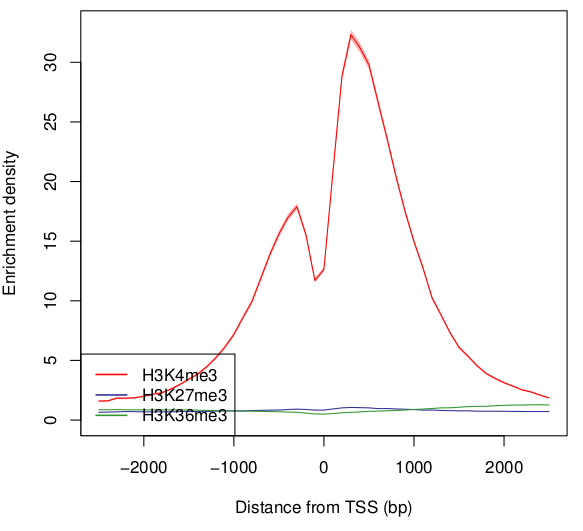
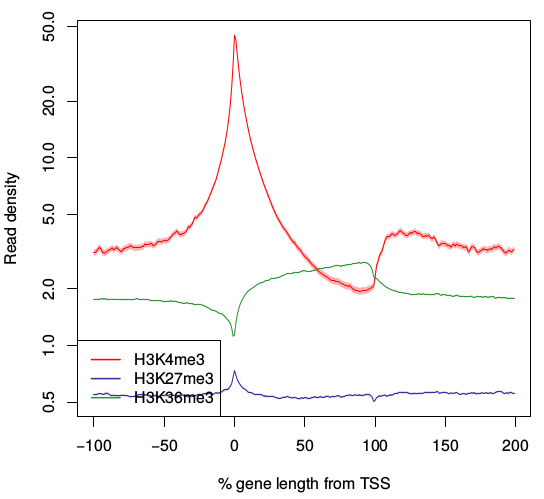
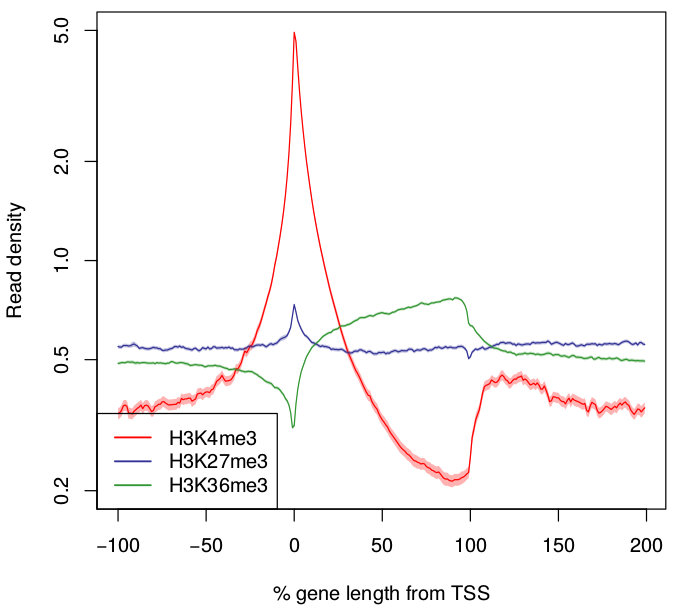
せっかくNature Protocolの論文があるので試してみよう企画。
Nature Protocolはスクリプトがそのまま載っているので、追試に最適です。
一方、古いライブラリが指定されていると手元の環境で動かなかったり、著者のレベルによってへんてこなスクリプトになっていたりしますが…
今回使うのは以下の論文です。
HISAT-StringTie-Ballgown については前回の記事を参照してください。
RNA-seqによる発現量解析 - Palmsonntagmorgen
hisat2, stringtieをインストール
hisat2, stringtie およびgffcompareも必要になりますので、コンパイル済ファイルをダウンロード、解凍します。
$ wget http://ccb.jhu.edu/software/hisat2/dl/hisat2-2.1.0-Linux_x86_64.zip $ wget http://ccb.jhu.edu/software/stringtie/dl/stringtie-1.3.5.Linux_x86_64.tar.gz $ wget http://ccb.jhu.edu/software/stringtie/dl/gffcompare-0.10.6.Linux_x86_64.tar.gz $ unzip hisat2-2.1.0-Linux_x86_64.zip $ tar zxvf stringtie-1.3.5.Linux_x86_64.tar.gz $ tar zxvf gffcompare-0.10.6.Linux_x86_64.tar.gz
解凍したらパスを通します。 ~/.barhrcに以下のような感じで記述し(場所は自分のディレクトリに合わせる)、source ~/.barhrcを実行します。
export PATH=$PATH:$HOME/hisat2-2.1.0/:$HOME/stringtie-1.3.5.Linux_x86_64:$HOME/gffcompare-0.10.6.Linux_x86_64/
パスが通ったか確認しましょう。
$ hisat2 --version /home/rnakato/git/binaries/hisat2-2.1.0/hisat2-align-s version 2.1.0 64-bit Built on login-node03 Wed Jun 7 15:53:42 EDT 2017 Compiler: gcc version 4.8.2 (GCC) Options: -O3 -m64 -msse2 -funroll-loops -g3 -DPOPCNT_CAPABILITY Sizeof {int, long, long long, void*, size_t, off_t}: {4, 8, 8, 8, 8, 8} $ stringtie --version 1.3.5 $ gffcompare --version gffcompare v0.10.6
バージョンが返ってきたら成功です。Not foundであればパスが通っていませんので.bashrcを見直しましょう。
Ballgownのインストール
BallgownはRで動きますので、R上で以下のコマンドを実行してインストールします。
> install.packages("devtools",repos="http://cran.us.r-project.org")
> source("http://www.bioconductor.org/biocLite.R")
> biocLite(c("alyssafrazee/RSkittleBrewer","ballgown","genefilter","dplyr","devtools"))
上記コマンドはNature protocolそのままですが、ballgownと共に必要になる関連ツール群もインストールされているようです。
最初にdevtoolsをインストールしていますが、これはgithub上のツールをインストールするためのツールです。
biocLiteはBioconductorからツールをインストールするコマンドで、sourceコマンドで参照元URLを指定します。
データダウンロード
次に使用するデータ群をダウンロードします。これは論文で提供されているものです。
$ wget ftp://ftp.ccb.jhu.edu/pub/RNAseq_protocol/chrX_data.tar.gz $ wget ftp://ftp.ccb.jhu.edu/pub/RNAseq_protocol/hg38_ucsc.annotated.gtf $ tar zxvf chrX_data.tar.gz
これはけっこう時間がかかります。
解凍されたディレクトリに何が入っているか確認しましょう。
$ ls chrX_data/*
chrX_data/geuvadis_phenodata.csv chrX_data/mergelist.txt
chrX_data/genes:
chrX.gtf
chrX_data/genome:
chrX.fa
chrX_data/indexes:
chrX_tran.1.ht2 chrX_tran.3.ht2 chrX_tran.5.ht2 chrX_tran.7.ht2
chrX_tran.2.ht2 chrX_tran.4.ht2 chrX_tran.6.ht2 chrX_tran.8.ht2
chrX_data/samples:
ERR188044_chrX_1.fastq.gz ERR188257_chrX_1.fastq.gz ERR188401_chrX_1.fastq.gz
ERR188044_chrX_2.fastq.gz ERR188257_chrX_2.fastq.gz ERR188401_chrX_2.fastq.gz
ERR188104_chrX_1.fastq.gz ERR188273_chrX_1.fastq.gz ERR188428_chrX_1.fastq.gz
ERR188104_chrX_2.fastq.gz ERR188273_chrX_2.fastq.gz ERR188428_chrX_2.fastq.gz
ERR188234_chrX_1.fastq.gz ERR188337_chrX_1.fastq.gz ERR188454_chrX_1.fastq.gz
ERR188234_chrX_2.fastq.gz ERR188337_chrX_2.fastq.gz ERR188454_chrX_2.fastq.gz
ERR188245_chrX_1.fastq.gz ERR188383_chrX_1.fastq.gz ERR204916_chrX_1.fastq.gz
ERR188245_chrX_2.fastq.gz ERR188383_chrX_2.fastq.gz ERR204916_chrX_2.fastq.gz
ゲノム配列や遺伝子ファイル、hisat2用のインデックス、fastqデータなどが入っています。 ファイルサイズと計算量を抑えるために、chrXだけを抽出したもののようですね。
マッピング
ツールとデータが揃いましたので、早速実験しましょう。まずはhisat2によるマッピングです。
for prefix in ERR188044 ERR188104 ERR188234 ERR188245 ERR188257 ERR188273 ERR188337 ERR188383 ERR188401 ERR188428 ERR188454 ERR204916
do
hisat2 -p 16 --dta -x chrX_data/indexes/chrX_tran \
-1 chrX_data/samples/${prefix}_chrX_1.fastq.gz \
-2 chrX_data/samples/${prefix}_chrX_2.fastq.gz \
-S ${prefix}_chrX.sam
samtools sort -@ 16 -o ${prefix}_chrX.bam ${prefix}_chrX.sam
done
論文ではわかりやすさのため1サンプルずつ順番に書き下していますが、面倒なので、ここではfor文を使って全サンプルを順にマッピングしています。
${prefix} のところにERR188044, ERR188104, ... と順に入っていく感じです。
論文ではCPU数を8に指定していましたが、早く計算したいのでここでは16にしています。
ついでにsamtoolsによるsort.bamへの変換も同時に行っています。
手元の環境では1サンプルにつき数十秒で計算終了しました。
遺伝子ファイル作成、発現量計測
次にStringtieを使って発現量を計測します。こちらもfor文で回します。
for prefix in ERR188044 ERR188104 ERR188234 ERR188245 ERR188257 ERR188273 ERR188337 ERR188383 ERR188401 ERR188428 ERR188454 ERR204916 do stringtie -G chrX_data/genes/chrX.gtf -o ${prefix}_chrX.gtf \ -p 16 -l ${prefix} ${prefix}_chrX.bam done
入力がbamファイル、出力がgtfファイルです。 完了したら、各サンプルのgtfファイルをマージしましょう。
$ stringtie --merge -p 8 -G chrX_data/genes/chrX.gtf \ -o stringtie_merged.gtf chrX_data/mergelist.txt
入力となる各サンプルのgtfファイルは、chrX_data/mergelist.txt内に記載されています。
ここでは著者が親切にリストを既に作ってくれていますが、通常は自分で新規作成するものです。
遺伝子ファイルの比較
論文ではoptionalとなっていますが、gffcompareを使って、生成された遺伝子リストとreferenceの遺伝子リストを比較することができます。
$ gffcompare -r chrX_data/genes/chrX.gtf -G -o merged stringtie_merged.gtf
この作業により、referenceに含まれない新規転写物を探索することができます。
次回に続きます。